Caracterización Molecular de Mutaciones en Enzimas de la Cadena de Transporte de Electrones: Un Enfoque Integrado desde el Diseño de Primers hasta el Cálculo de la Temperatura de Fusión
Authors: María Azucena Jurado Estrada 1
1 Biotechnology career, Faculty of Science, Campus "El cerrillo" Universidad Autónoma del Estado de México.
Abstract:
Anteriormente se exploró la energía libre de Gibbs a lo largo de la respiración celular, desde la glucólisis hasta la cadena transportadora de electrones, indicando la favorabilidad de las reacciones bioquímicas que producen ATP. El objetivo de este artículo es a partir de la cadena transportadora de electrones, tomar las enzimas que tienen mutaciones en esta ruta bioquímica y con documentación entender dónde están las mutaciones de esas enzimas y mapear los exones donde se indique mutación y calcular el Tm con el artículo de Santa Lucia. Se analizaron las siguientes enzimas de la cadena transportadora de electrones: ubiquinol-cytochrome c reductase core protein 2,
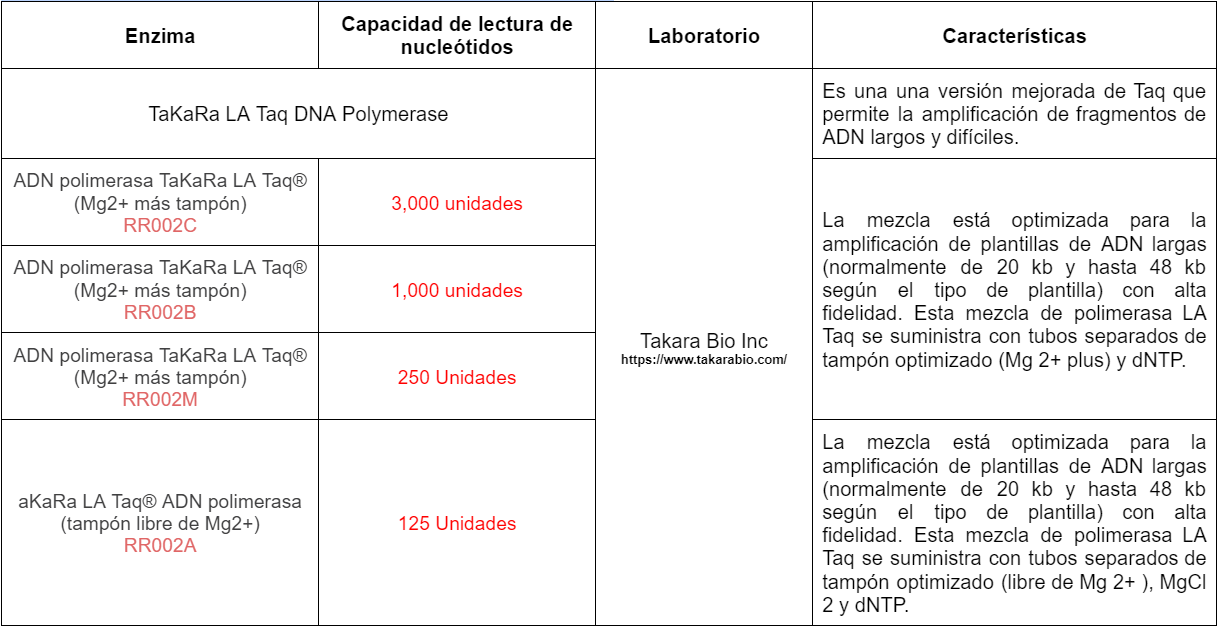
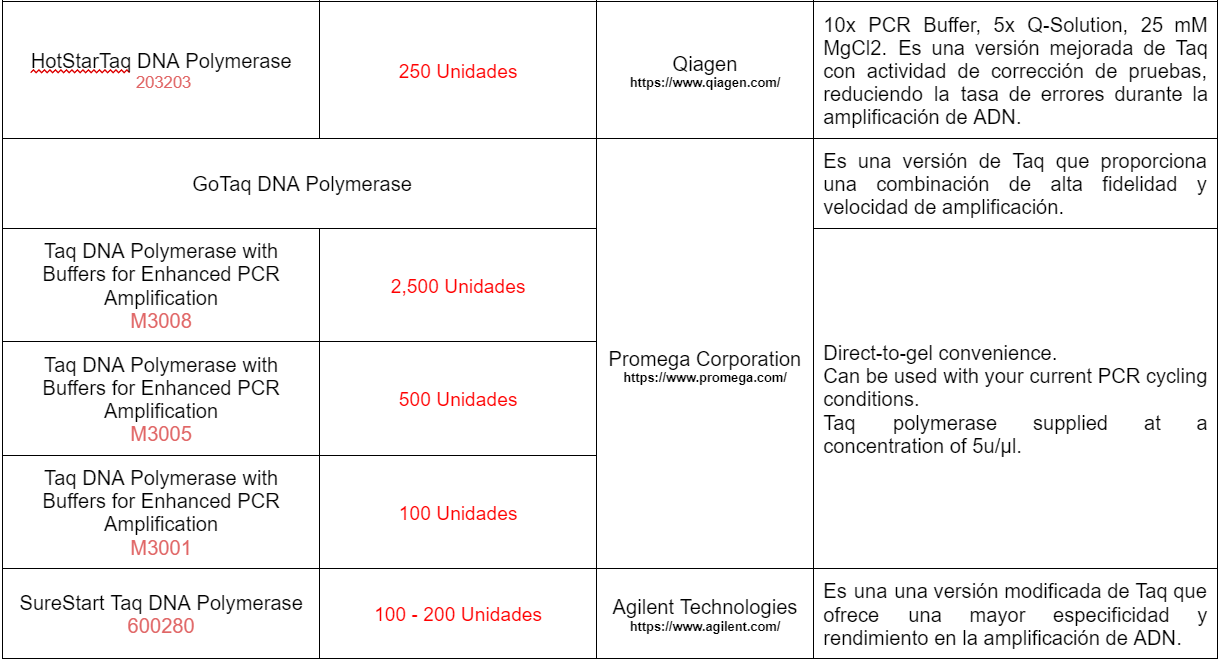
En el diseño de primers, comprender la entropía de una secuencia objetivo es necesaria para seleccionar regiones adecuadas y diseñar primers específicos y eficientes. Una baja entropía indica que la región es altamente conservada y por lo tanto, puede ser menos útil para el diseño de primers, mientras una alta entropía sugiere que la región es más variable, lo que puede ser útil para el diseño de primers específicos para una variante genética específica. Se hizo una búsqueda de enzimas para la PCR que permitir la amplificación selectiva y específica de secuencias de ADN objetivo seleccionando las enzimas ADN polimerasa TaKaRa LA Taq® (Mg2+ más tampón) del laboratorio Takara Bio Inc.
Introduction
Anteriormente se examinó en profundidad la respiración como un proceso multifacético que abarca mucho más que el simple intercambio de gases. Este proceso incluye la producción de energía y la contribución al mantenimiento de la homeostasis a través del ajuste del pH de los fluidos corporales. Se destacan dos conceptos fundamentales: la ventilación pulmonar y la respiración celular. La ventilación pulmonar implica la difusión del oxígeno inhalado en los pulmones a través de las membranas alveolares y capilares pulmonares hacia la sangre, donde se une a la hemoglobina en los glóbulos rojos para su transporte. Este oxígeno es esencial para la cadena de transporte de electrones en la respiración celular, donde actúa como aceptor de electrones y facilita la producción de ATP a partir de NADH y FADH2 generados en procesos metabólicos como la glucólisis y el ciclo de Krebs.
En cuanto a la respiración celular, se detallan las etapas clave: la glucólisis, la conversión de piruvato en acetil-CoA, el ciclo de Krebs y la cadena de transporte de electrones. Durante la glucólisis, la glucosa se descompone en el citoplasma celular para formar piruvato, produciendo ATP y NADH. El complejo enzimático de la piruvato deshidrogenasa, que convierte el piruvato en acetil-CoA dentro de la matriz mitocondrial, liberando CO2 y generando NADH. En el ciclo de Krebs, el acetil-CoA se oxida para producir NADH, FADH2 y GTP, y liberar CO2. Finalmente, en la cadena de transporte de electrones, NADH y FADH2 donan electrones que generan un gradiente electroquímico utilizado para sintetizar ATP a través de la fosforilación oxidativa, con el oxígeno actuando como el aceptor final de electrones y formando agua.
Se profundizó en los cambios en la energía libre de Gibbs a lo largo de las etapas de la respiración celular, desde la glucólisis hasta la cadena de transporte de electrones; como se estudió la energía libre de Gibbs es una medida crítica de la favorabilidad de las reacciones bioquímicas. Los resultaros que se obtuvieron en la sumatoria fueron los siguientes: La glucólisis tiene una energía libre de Gibbs de -73.3 kJ/mol; la conversión del piruvato a acetil-CoA tiene una energía libre de Gibbs de -33.4 kJ/mol; el ciclo de Krebs tiene una energía libre de Gibbs total de -114.5 kJ/mol, y la fosforilación oxidativa en la cadena de transporte de electrones tiene un valor altamente negativo de -220.1 kJ/mol. Estos valores al ser negativos, indicaron que son procesos altamente favorables que permiten a la célula realizar funciones vitales y mantener el metabolismo. Así mismo se observó la importancia de la favorabilidad de reacciones en la homeostasis del pH, especialmente en condiciones patológicas como la hiperventilación y la tuberculosis pulmonar, alterando la eficiencia metabólica celular Reyes, J. I. V., Estrada, M. A. J., & Jimenez., E. G. C. (2024).
Relación entre entropía y diseño de primers:
En el diseño de primers para la PCR, la entropía se utiliza como una medida de la incertidumbre o variabilidad presente en una secuencia de ADN específica. Shannon (1948) establece que la entropía es una medida de la cantidad de información necesaria para describir o predecir un evento. En el contexto del ADN, una secuencia altamente conservada, es decir, una secuencia que muestra una alta homología o similitud con otras secuencias, tendrá una entropía baja. Esto se debe a que una secuencia conservada contiene poca información nueva o variabilidad, lo que significa que es más predecible y menos incierta.
Investigaciones recientes han explorado cómo la entropía puede integrarse en los procesos de diseño de primers para mejorar la eficiencia y la especificidad de la amplificación.Gu et al. (2010), utilizó la entropía para evaluar la variabilidad de secuencias de ADN en diferentes regiones genómicas. Los autores encontraron que las regiones altamente conservadas tenían una entropía baja, mientras que las regiones más variables mostraban una entropía más alta.
Por otro lado, una secuencia más variable, que exhibe una mayor diversidad en sus nucleótidos o presenta mutaciones frecuentes, tendrá una entropía más alta. Esto se debe a que una secuencia variable contiene más información nueva o impredecible, lo que aumenta la incertidumbre asociada con la secuencia; por lo que la entropía de una secuencia de ADN refleja la cantidad de variabilidad presente en esa secuencia en particular Chen et al. (2008).
Diseño de primers y la PCR:
Los primers sirven como iniciadores para la amplificación selectiva de la región de interés durante la Reacción en Cadena de la Polimerasa (PCR), siendo una técnica que ayuda a la amplificación in vitro de secuencias de ADN específicas Mullis et al., (1986). Los primers son diseñados para emparejarse con regiones flanqueantes a ambos lados de la secuencia de interés, permitiendo así que la ADN polimerasa sintetice nuevas hebras de ADN complementarias a partir de la plantilla de ADN original (Saiki et al., 1988). Este proceso de amplificación permite la detección, cuantificación y análisis de secuencias de ADN específicas, lo que tiene aplicaciones en una amplia variedad de campos(Untergasser et al., 2012).
Es importante considerar el diseño de primers, la temperatura de fusión (Tm), el contenido de GC y la selección de enzimas para la reacción en cadena de la polimerasa (PCR) para lograr resultados precisos y eficientes en las reacciones de PCR, existen diferentes enzimas que poseen características particulares que pueden influir en la eficiencia y fidelidad de la amplificación, un ejemplo son Taq DNA polimerasa, son las utilizadas debido a su estabilidad a altas temperaturas, sin embargo, enzimas con actividad de corrección de pruebas, como Pfu DNA polimerasa, son preferidas en aplicaciones donde la alta fidelidad es esencial" (Innis, M.A., & Gelfand, D.H. (1990).
A continuación se muestran alteraciones genéticas en enzimas de la fosforilación oxidativa.
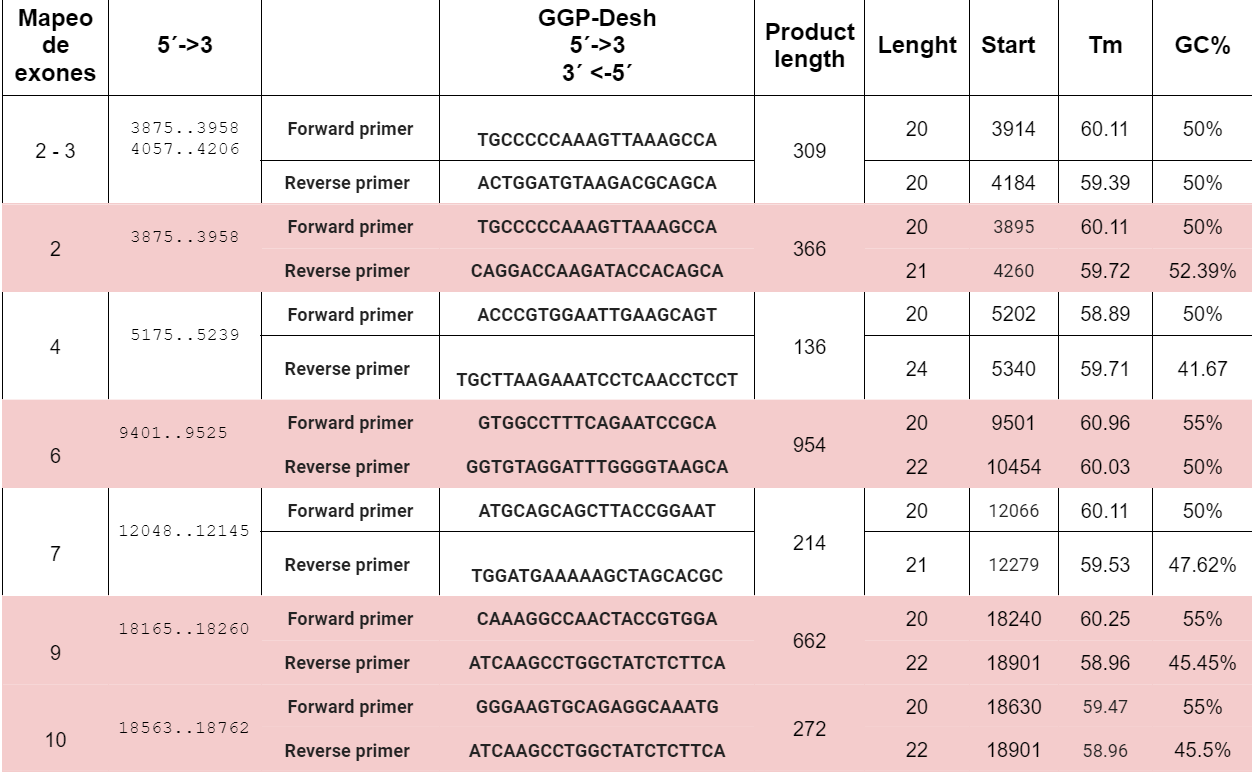
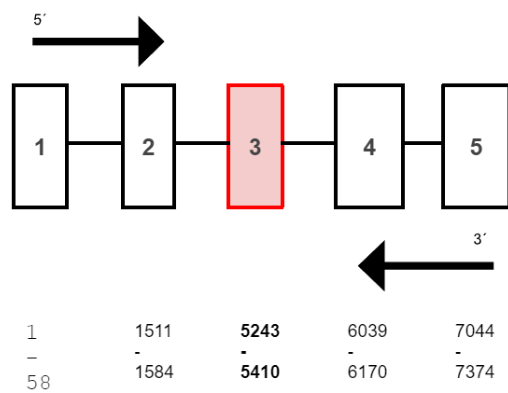
- Homo sapiens ubiquinol-cytochrome c reductase core protein 2 (UQCRC2), RefSeqGene on chromosome 16; nuclear gene for mitochondrial product
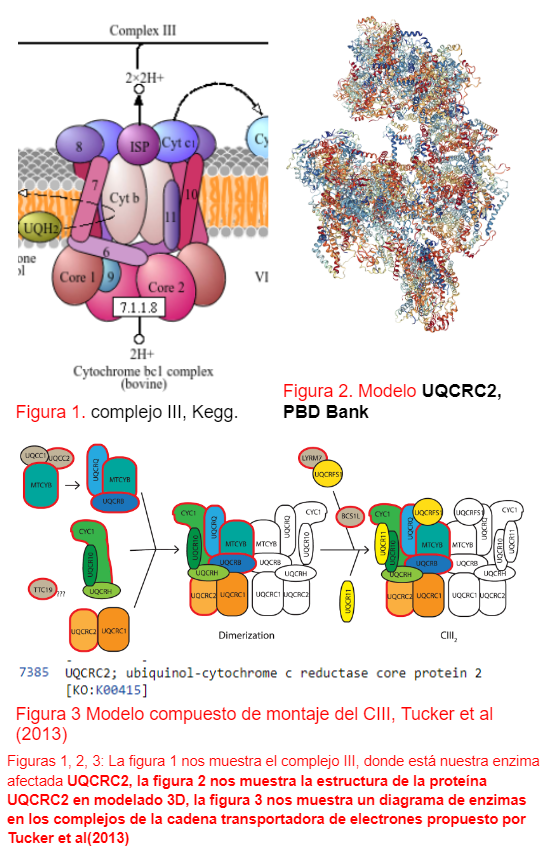
Es una subunidad esencial del Complejo III de la cadena transportadora de electrones en la mitocondria. Localizado en la membrana interna mitocondrial, desempeña un papel crítico en la transferencia de electrones desde el ubiquinol al citocromo c. También oxida la ubiquinol a ubiquinona y reduce dos moléculas de citocromo c, lo que contribuye a la creación de un gradiente de protones mediante el bombeo de protones desde la matriz mitocondrial al espacio intermembrana. Este gradiente es fundamental para la síntesis de ATP en el Complejo V (ATP sintasa).
Alteraciones genéticas que puede Inhibir el funcionamiento
Las mutaciones en el gen UQCRC2 pueden alterar la estructura y función del Complejo III, resultando en una deficiencia en la transferencia de electrones, producción insuficiente de ATP y acumulación de especies reactivas de oxígeno (ROS), lo cual puede causar daño celular y contribuir a enfermedades mitocondriales. Un estudio realizado por (Ait-El-Mkadem y colaboradores en 2017) examinó las mutaciones en genes mitocondriales en pacientes con enfermedades mitocondriales. Encontraron que las mutaciones en UQCRC2 estaban presentes en varios exones del gen, incluyendo el exón 2 y el exón 9. Estas mutaciones afectaban la estructura y función del Complejo III de la cadena transportadora de electrones, lo que resultaba en una disfunción mitocondrial y una variedad de manifestaciones clínicas en los pacientes.
Además, un estudio más reciente realizado por (Martínez-Reyes y colaboradores en 2021) investigó las mutaciones en UQCRC2 en relación con la hipertensión pulmonar asociada a la enfermedad de células falciformes. Encontraron que las mutaciones en el exón 6 y el exón 10 del gen UQCRC2 estaban asociadas con un aumento en la producción de ROS y disfunción endotelial en los pacientes, lo que contribuía al desarrollo de hipertensión pulmonar.
2. Citocromo c oxidasa (complejo IV):
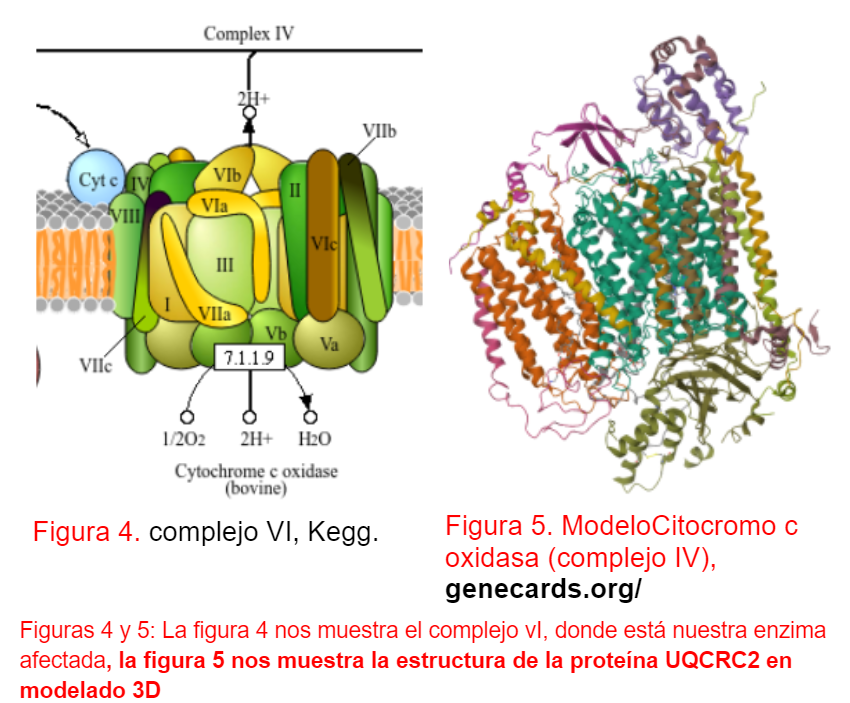
La citocromo c oxidasa es un complejo multimerico de proteínas localizado en la membrana mitocondrial interna, es esencial para la última etapa de la cadena de transporte de electrones, donde cataliza la reducción del oxígeno molecular a agua. Codificado por genes como MT-CO1 en el ADN mitocondrial, este complejo enzimático realiza una función crítica en el flujo de electrones y la generación de un gradiente electroquímico necesario para la síntesis de ATP, se muestra una alteración en el exón 3, las mutaciones en el gen MT-CO1 pueden afectar la estructura o la función de la citocromo c oxidasa, lo que resulta en una disminución en la capacidad del complejo IV para reducir el oxígeno y, por lo tanto, una reducción en la producción de ATP DiMauro S, Schon EA, Carelli V, Hirano M. Muscle Nerve 2006.
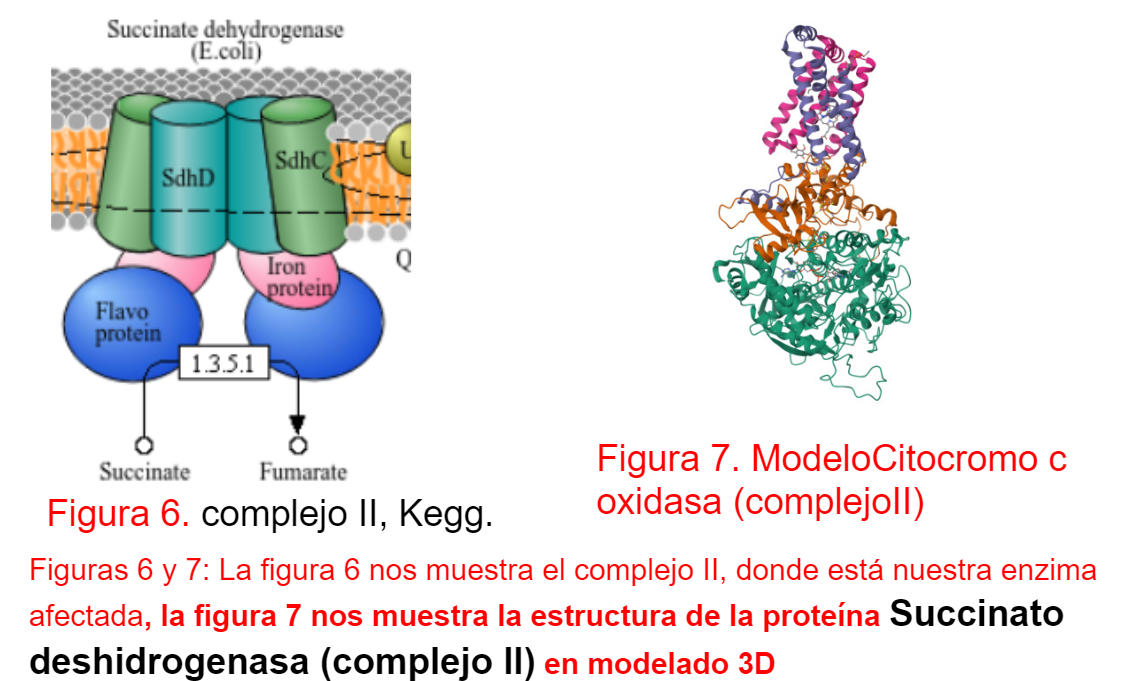
- Succinato deshidrogenasa (complejo II):
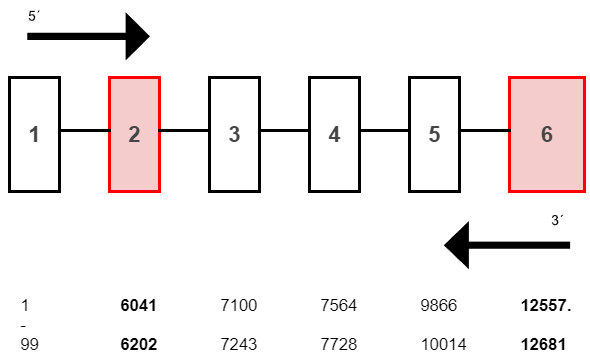
La succinato deshidrogenasa está codificada por los genes SDHA, SDHB, SDHC y SDHD, los cuales se encuentran en el ADN nuclear, acorde a la bibliografía el gen SDHB, tiene mutaciones en los exones 2, 6. Acorde a la literatura la falla en este gen conduce a una disminución en la actividad del complejo II y, por lo tanto, a una reducción en la generación de energía durante la respiración celular Jansen JC, van den Berg R, Kuiper A, et al. Clin Genet. 2006.
El objetivo de este artículo es a partir de la cadena transportadora de electrones, se analizaron las enzimas que tienen mutaciones de los complejos:UQCRC2 y con documentación se analizó dónde están las mutaciones de esas enzimas, se mapearon los exones donde se indica la mutación, así mismo se hace un análisis comparativo de las fórmulas para el cálculo del Tm , Promega, fórmula 5 del artículo de Santa Lucía y de NCBI.
Materials and methods:
Se utilizó NCBI Primer-BLAST, que es una herramienta en línea proporcionada por el National Center for Biotechnology Information (NCBI) que permite buscar primers específicos para amplificar secuencias de ADN. Se ingresaron varias secuencias de interés enfocadas a las enzimas de la cadena transportadora de electrones, a través de documentación de se buscaron alteraciones genéticas de estas enzima; esta herramienta nos ayudó a identificar regiones de interés acorde a la documentación para el diseño de primers específicos.
Se hizo una búsqueda de enzimas para la PCR que permitir la amplificación selectiva y específica de secuencias de ADN objetivo, se buscaron en laboratorios como: Thermo Fisher Scientific; New England Biolabs; Qiagen; Promega Corporation; Takara Bio Inc;
Agilent Technologies; Bio-Rad Laboratories (revisar anexo 1), por lo que en el diseño de primers se cambió la configuración de 3000 mucleótidos de lectura de NCBI , como capacidad de lectura de nucleótidos seleccionando las enzimas ADN polimerasa TaKaRa LA Taq® (Mg2+ más tampón) Takara Bio Inc. Se procedió a utilizar Swiss Model, para el modelado de proteínas y kegg para la ubicación visual de la proteína, se añadió la fórmula 5 del artículo de McTigue,P (2004). Se realizó un análisis comparativo de las fórmulas para el cálculo del Tm , Promega, fórmula 5 del artículo de Santa Lucía y de NCBI.
Results:
- Pathway: Homo sapiens ubiquinol-cytochrome c reductase core protein 2 (UQCRC2), RefSeqGene on chromosome 16; nuclear gene for mitochondrial product
Exones afectados:2,6,9,10

- Homo sapiens cytochrome c oxidase subunit 4I1 (COX4I1), RefSeqGene on chromosome 16; nuclear gene for mitochondrial product
Exones afectados: Exón 3

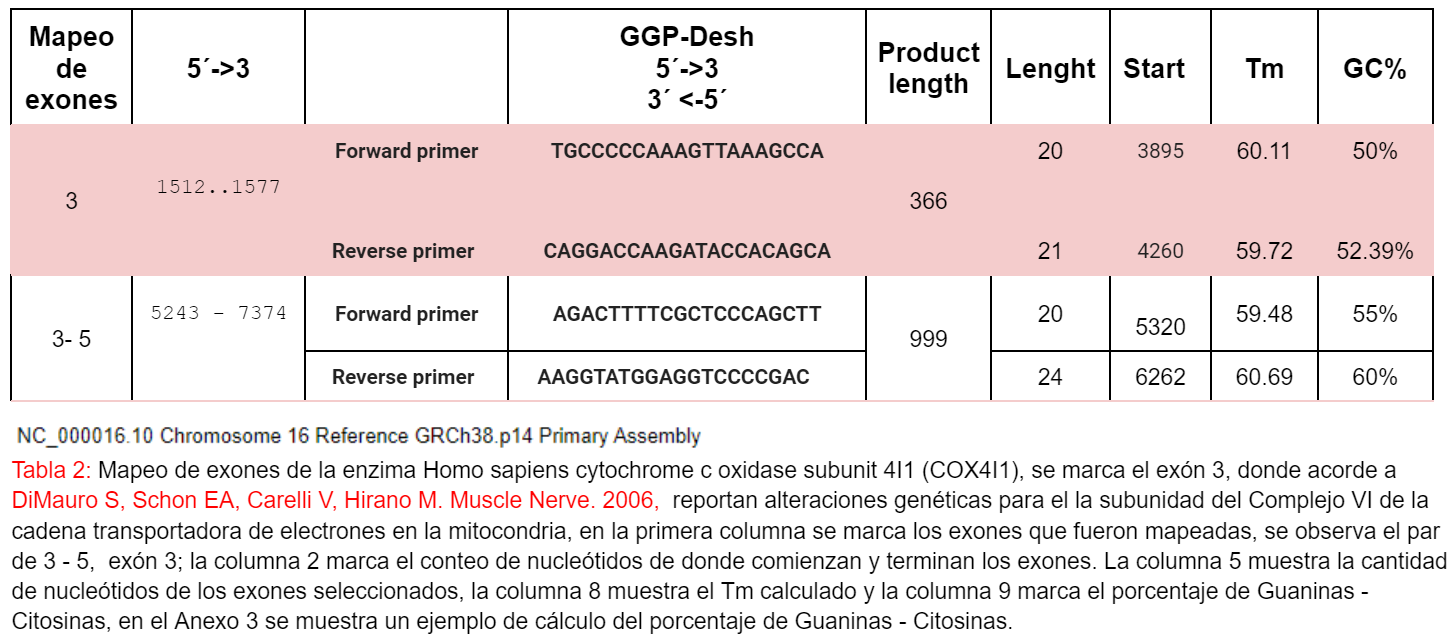
- Succinato deshidrogenasa (complejo II):
Exones afectados: Exones 2, 6

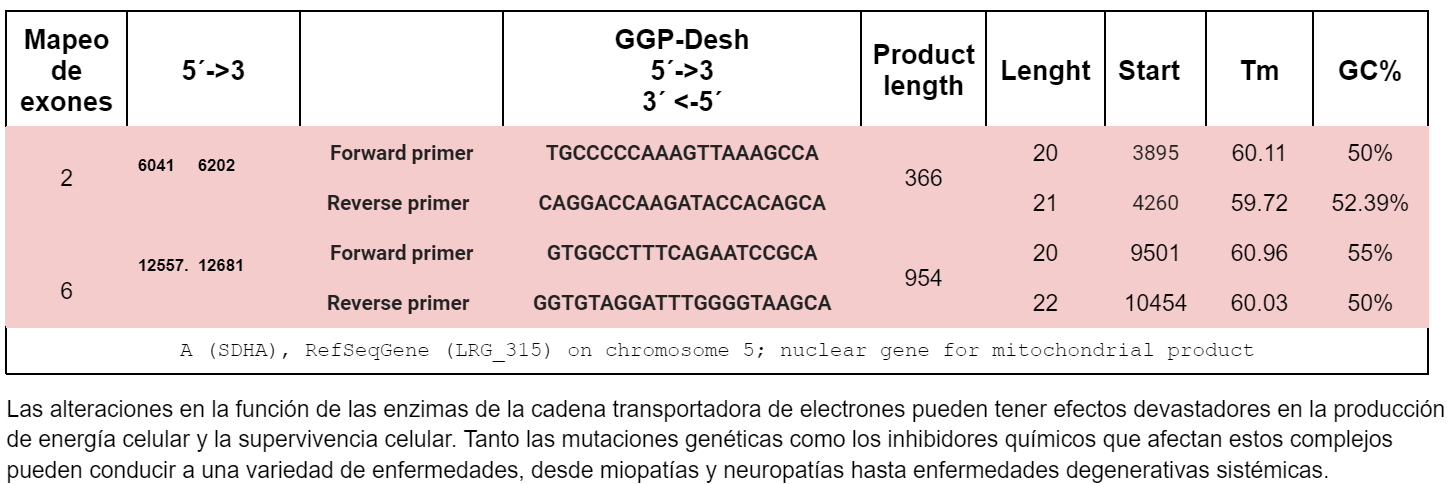
Análisis comparativo de las fórmulas para el cálculo del Tm , Promega, fórmula 5 del artículo de Santa Lucía y de NCBI.



Discusión:
La identificación detallada de mutaciones en los exones específicos de las enzimas de la cadena transportadora de electrones tiene implicaciones significativas para el diagnóstico y tratamiento de enfermedades mitocondriales. Por ejemplo, las mutaciones en los exones 2, 6, 9 y 10 del gen UQCRC2 están vinculadas a enfermedades mitocondriales y hipertensión pulmonar. Estos hallazgos permiten una mejor comprensión de las bases moleculares de estas patologías y podrían conducir al desarrollo de terapias dirigidas que corrijan o compensen las deficiencias en la cadena transportadora de electrones. El uso de nuevas tecnologías y enfoques en el diseño de primers, como el análisis de entropía y herramientas avanzadas como NCBI Primer-BLAST, ha demostrado ser efectivo para mejorar la especificidad y eficiencia de la PCR. El enfoque en regiones de alta entropía para el diseño de primers permite una mayor precisión en la amplificación de secuencias genéticas específicas, especialmente aquellas con variantes genéticas importantes. La utilización de fórmulas precisas para el cálculo de Tm, como las proporcionadas por Promega, Santa Lucía y NCBI, optimiza las condiciones de PCR, garantizando resultados más fiables y reproducibles. Estos avances tienen un impacto considerable en la investigación genética y la práctica clínica. La capacidad de diseñar primers específicos y eficientes facilita la detección y análisis de mutaciones genéticas, mejorando el diagnóstico de enfermedades genéticas y mitocondriales. Además, la identificación de mutaciones en exones específicos y su relación con patologías proporciona una base sólida para estudios futuros y el desarrollo de tratamientos personalizados.
Futuras Direcciones:Expansión del Análisis Genético: Ampliar el análisis a otras enzimas dentro de la cadena transportadora de electrones y otros componentes críticos del metabolismo mitocondrial para identificar mutaciones adicionales y comprender mejor sus implicaciones clínicas.
Desarrollo de Terapias Dirigidas:Hay mucho que Investigar y desarrollar terapias que aborden directamente las mutaciones identificadas en genes como UQCRC2, MT-CO1 y SDHB, potencialmente mediante enfoques de edición genética o terapias génicas.
Mejora de Herramientas Bioinformáticas: Se debe continuar desarrollando y refinando herramientas bioinformáticas que integren el análisis de entropía y otros parámetros relevantes para el diseño de primers, mejorando la precisión y eficiencia de la PCR y otras técnicas de amplificación de ADN.
Estudios Clínicos y Epidemiológicos: Se pueden realizar estudios clínicos y epidemiológicos para correlacionar las mutaciones genéticas específicas con las manifestaciones clínicas y la progresión de las enfermedades, proporcionando una comprensión más profunda de la fisiopatología mitocondrial y sus variaciones individuales.
Acknowledgements
Thanks to Dr. Ricardo Lara, who has been with us since the beginning of our career, has shared with us his passion for science and has helped us develop our personality as students and future scientists. Thank you for your advice and for helping us discover that discipline and focus are an important basis for research, as well as for providing us with the technological tools and teaching us how to use them to do this work.
Conclusiones:
Este estudio proporciona una visión integral de la importancia de las mutaciones en los exones específicos de las enzimas de la cadena transportadora de electrones y su impacto en enfermedades mitocondriales, subrayando la relevancia de los exones 2, 6, 9 y 10 del gen UQCRC2 en patologías como la hipertensión pulmonar. Además, destaca la eficacia de nuevas tecnologías y enfoques en el diseño de primers, como el análisis de entropía y el uso de herramientas avanzadas como NCBI Primer-BLAST, que mejoran la especificidad y eficiencia de la PCR. Estos hallazgos no solo mejoran la comprensión de las bases moleculares de las enfermedades mitocondriales, sino que también ofrecen una base sólida para futuras investigaciones y el desarrollo de terapias personalizadas, enfatizando la necesidad de seguir avanzando en el análisis genético, el desarrollo de terapias dirigidas y la mejora de herramientas bioinformáticas.
Referencias
Introducción
Reyes, J. I. V., Estrada, M. A. J., & Jimenez., E. G. C. (2024). Clinical respiratory alterations and their biochemical implications and thermodynamic parameters in cellular respiration. Biotechnology career, Faculty of Science, Campus “El cerrillo” Universidad Autónoma del Estado de México.
Shannon, C. E. (1948). A Mathematical Theory of Communication. The Bell System Technical Journal, 27(3), 379–423.
Gu, J. et al. (2010). Sequence entropy-variability plots based on alignments of very large numbers of sequences can be used to extract protein modulators of functional activity. Protein Engineering Design and Selection, 23(9), 313–320.
Chen, J., Deng, Y., Luo, Q., & Liu, L. (2008). Primer design assistant (PDA): a web-based primer design tool. Nucleic Acids Research, 36(Web Server issue), W265–W269.
Mullis, K., Faloona, F., Scharf, S., Saiki, R., Horn, G., & Erlich, H. (1986). Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harbor Symposia on Quantitative Biology, 51(Pt 1), 263-273.
Saiki, R. K., Gelfand, D. H., Stoffel, S., Scharf, S. J., Higuchi, R., Horn, G. T., Mullis, K. B. (1988). Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science, 239(4839), 487-491.
Untergasser, A., Cutcutache, I., Koressaar, T., Ye, J., Faircloth, B. C., Remm, M., & Rozen, S. G. (2012). Primer3: new capabilities and interfaces. Nucleic Acids Research, 40(15), e115.
DiMauro S, Schon EA, Carelli V, Hirano M. Muscle Nerve. 2006 Clinical and molecular features of mitochondrial myopathies. Nov;34(5):587-605. DOI: 10.1002/mus.20615.
Resultados:
Ait-El-Mkadem, S., Dayem-Quere, M., Gusic, M., Chaussenot, A., Bannwarth, S., François, B & Abi Warde, M. T. (2017). Mutations in MDH2, encoding a Krebs cycle enzyme, cause early-onset severe encephalopathy. American Journal of Human Genetics, 100(1), 151-159.
Martínez-Reyes, I., Baban, B., Billoud, B., Papai, G., Sadek, M., Casola, C. & Jain, M. (2021). Disruption of the UQCRC2 subunit of mitochondrial complex III increases reactive oxygen species production and causes endothelial dysfunction in humans and mice. Circulation Research, 128(10), 1289-1306.
Saraste, M. (1999). Oxidative phosphorylation at the fin de siècle. Science, 283(5407), 1488-1493.
Hunte, C., Koepke, J., Lange, C., Rossmanith, T., & Michel, H. (2000). Structure at 2.3 A resolution of the cytochrome bc1 complex from the yeast Saccharomyces cerevisiae co-crystallized with an antibody Fv fragment. Structure, 8(6), 669-684.
Schapira, A. H. V. (2012). Mitochondrial diseases. The Lancet, 379(9828), 1825-1834.
Trumpower, B. L. (1990). The protonmotive Q cycle: Energy transduction by coupling of proton translocation to electron transfer by the cytochrome bc1 complex. Journal of Biological Chemistry, 265(20), 11409-11412.
Murphy, M. P. (2009). How mitochondria produce reactive oxygen species. Biochemical Journal, 417(1), 1-13.
Fórmulas:
SantaLucia, J. (2004). "The thermodynamics of DNA structural motifs." Annual Review of Biophysics and Biomolecular Structure, 33, 415-440.
Sugimoto, N., Nakano, S., Yoneyama, M., & Honda, K. (1996). "Improved thermodynamic parameters and helix initiation factor to predict stability of DNA duplexes." Nucleic Acids Research, 24(22), 4501-4505.
Breslauer, K. J., Frank, R., Blöcker, H., & Marky, L. A. (1986). "Predicting DNA duplex stability from the base sequence." Proceedings of the National Academy of Sciences, 83(11), 3746-3750.
Von Ahsen, N., Wittwer, C. T., & Schütz, E. (2001). "Oligonucleotide melting temperatures under PCR conditions: nearest-neighbor corrections for Mg2+, deoxynucleotide triphosphate, and dimethyl sulfoxide concentrations with comparison to alternative empirical formulas." Clinical Chemistry, 47(11), 1956-1961.
Búsqueda de enzimas para la PCR
Agilent Technologies. (s/f). SureStart Taq DNA Polymerase. Agilent Technologies. Recuperado el 31 de mayo de 2024, de https://www.agilent.com/
Bio-Rad Laboratories. (s/f). iProof High-Fidelity DNA Polymerase. Bio-Rad Laboratories. Recuperado el 31 de mayo de 2024, de https://www.bio-rad.com/es-mx
New England Biolabs (NEB). (s/f). Phusion® High-Fidelity DNA Polymerase. New England Biolabs (NEB). Recuperado el 31 de mayo de 2024, de https://www.neb.com/
Promega Corporation. (s/f). GoTaq DNA Polymerase. Promega Corporation. Recuperado el 31 de mayo de 2024, de https://www.promega.com/
Qiagen. (s/f). HotStarTaq DNA Polymerase. Qiagen. Recuperado el 5 de 2024, de https://www.qiagen.com/
Scientific, T. F. (s/f). Platinum II Taq Hot-Start DNA Polymerase; AmpliTaq Gold 360 DNA Polymerase; Phusion DNA Polymerase. Thermo Fisher Scientific. Recuperado el 31 de mayo de 2024, de https://www.thermofisher.com/
Takara Bio Inc. (s/f). TaKaRa LA Taq DNA Polymerase. Takara Bio Inc. Recuperado el 31 de mayo de 2024, de
Anexo 1
Búsqueda de enzimas para la PCR